درخت تبارزایی - ویکیپدیا، دانشنامهٔ آزاد

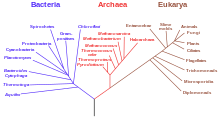
درخت تبارزایی، درخت فیلوژنتیک، درخت تکامل نژادی یا درخت تکاملی (به انگلیسی:Phylogenetic tree) یک نمودار انشعابی است (که اصطلاحاً درخت (نظریه گراف) نامیده میشود) و روابط تکاملی در میان گونههای مختلف زیستی یا حتی اشخاص را بر اساس شباهتها و تفاوتهای فیزیکی (تبارزایی) یا خصوصیات ژنتیکی نشان میدهد. واحدهایی که به هم در درخت متصل هستند، از یک نسب مشترک جدا شدهاند. در یک درخت تکامل نژادی ریشهدار، هر گره نشاندهندهٔ نسب مشترکی برای فرزندان آن گره است و طول یالها در برخی از درختان نشاندهندهٔ تخمین زمان است. هر گره یک واحد آرایهای نامیده میشود. گرههای داخلی عموماً واحد طبقهبندی فرضی (HTUs) نامیده میشوند و نمیتوان آنها را بهطور مستقیم مشاهده کرد. این درختان در زمینههای زیستشناسی مانند زیستشناسی فرگشتی، بیوانفورماتیک، سیستماتیک و فیلوژنتیک مقایسهای کاربرد دارند.
تاریخچه[ویرایش]
ایدهٔ درخت زندگانی از متنهای باستانی، از یک فرایند نردبانی از پایینترین تا بالاترین نوع حیات (مانند یک زنجیر آفرینش) گرفته شدهاست. اولین درختهای منشعبکنندهٔ تبارزایی شامل یک نمودار دیرینهشناسی میشد که ارتباطات زمینشناسی میان گیاهان و جانوران را نشان میداد و در کتاب «زمینشناسی مقدماتی» نوشتهٔ ادوارد هیچکاک (چاپ اول ۱۸۴۰) آمده بود.
چارلز داروین یکی از اولین و محبوبترین تصاویر از مفهوم درخت تکامل را در کتاب اصلی خود، «خاستگاه گونهها» به تصویر کشیدهاست. در طول قرن بعد، زیستشناسی فرگشتی هنوز از نمودار درختی برای نمایش فرگشت استفاده میکردند، زیرا این نمودارها بهخوبی مفهوم گونهزایی را بیان میکنند. در طول زمان، دستهبندی گونهها کمتر ایستا و بیشتر پویا شد.
گونهها[ویرایش]
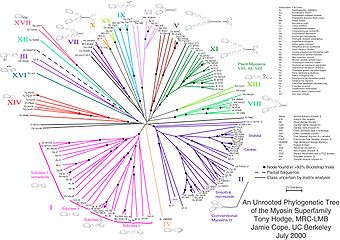
درخت فیلوژنتیک ریشهدار یک گراف جهتدار است با یک گره منحصر به فرد متناظر با نزدیکترین جد مشترک از همه جاندار که در برگهای درخت هستند. روش معمول ریشهدار کردن درخت استفاده از outgroup است. درختان بدون ریشه ارتباط گرههای برگ را بدون هیچ فرضی در مورد اصل و نسبشان نشان میدهند. در حالیکه درختان بدون ریشه به سادگی با حذف ریشهها از درختان ریشهدار تولید میشوند، از یک درخت بدون ریشه نمیتوان بدون استفاده از شناسایی اصل و نسب، ریشه را نتیجه گرفت. بهطور معمول ریشهدار کردن یک درخت با اضافه کردن outgroup در دادههای ورودی یا معرفی مفروضات بیشتری در مورد انجام سرعت نسبی تکامل در هر شاخه، مانند فرضیه ساعت مولکولی انجام میشود. شکل ۱ یک درخت تکامل نژادی بدون ریشه را برای پروتئین اصلی عضله نشان میدهد.
هر دو نوع درختان تکامل نژادی ریشهدار و بدون ریشه میتوانند یک قسمتی (bifurcating) یا چند قسمتی (multifurcating)، یا برچسبدار و بدون برچسب باشند. هر یک از گرههای داخلی در یک درخت یکقسمتی ریشهدار دقیقاً دارای دو فرزند است (شبیه یک درخت دوقسمتی)، و یک درخت یکقسمتی بدون ریشه یک درخت دوقسمتی است ولی هر گرهٔ داخلی دقیقاً سه همسایه دارد. در مقابل، یک درخت چندقسمتی ریشهدار ممکن است بیشتر از دو فرزند در برخی از گرهها داشته باشد و یک درخت چندقسمتی بدون ریشه ممکن است بیش از سه همسایه در برخی از گرهها داشته باشد. در یک درخت برچسبدار به هر یک از برگها ارزشهای خاصی اختصاص مییابد، در حالیکه یک درخت بدون برچسب فقط یک توپولوژی را تعریف میکند. با داشتن تعدادی گره برگ بسته به نوع خاص درخت تعدادی درخت وجود دارد، اما همیشه درختان چندقسمتی از درختان یکقسمتی بیشتر هستند و درختان برچسبدار بیشتر از درختان بدون برچسب و درختان ریشهدار بیشتر از درختان بدون ریشه وجود دارند. آخرین تمایز، بیشترین ارتباط بیولوژیکی را دارد، زیرا مکانهای بسیاری در یک درخت بدون ریشه برای قرار دادن ریشه وجود دارد.
برای درختان یکقسمتی برچسبدار به تعداد زیر، درخت ریشهدار وجود دارد:
و به تعداد زیر، درخت بدون ریشه وجود دارد:
که n تعداد برگهای درخت را مشخص میکند. در میان درختان دوتایی برچسبدار، تعداد درختان بدون ریشه با n برگ برابر تعداد درختان ریشهدار با n-1 برگ است.[۴]
اجزای درخت فیلوژنتیک[ویرایش]
درخت فیلوژنتیک وجود و انشعاب شاخهها و دودمانها را در طول زمان نشان میدهد. طول هر شاخه ممکن است اختیاری، یا برگرفته شده از مقیاس زمانی خاصی باشد که زمان بین وقوع گونهزاییها را نشان میدهد. نقطههایی را که در آنها دودمانها واگرا میشوند گره مینامند. هر گره نمایندهٔ یک نسب مشترک برای گونههای واگرا شونده در گره است. نوک شاخهها، نمایانگر گونهها، یا گروههای بزرگتری است که امروزه زنده اند، یا منقرض شدهاند. ریشه درخت فیلوژنتیک نشاندهندهٔ قدیمیترین نسب مشترک همة گروههای نشان دادهشده روی درخت است.[۵]
انواع درختان فیلوژنتیک[ویرایش]
- دندروگرام (dendrogram): اصطلاح گستردهای برای نمایش یک درخت تکامل نژادی است.
- کلادوگرام (cladogram): یک درخت تکامل نژادی است که با استفاده از روشهای کلادیستیک شکل گرفتهاست. این نوع درختان تنها نشاندهندهٔ یک الگوی انشعاب هستند، به عنوان مثال طول شاخههای آن، زمان یا مقدار نسبی تغییر کاراکترها را نشان نمیدهد.
- فیلوگرام (phylogram): یک درخت فیلوژنتیک است که طول هر انشعاب، میزان تغییرات را نشان میدهد.
- کرونوگرام (chronogram): یک درخت فیلوژنتیک است که طول هر انشعاب مدت زمان میان دو گونه را نشان میدهد.
ساختن درخت فیلوژنتیک[ویرایش]
درختهای فیلوژنتیک به عنوان یک روش غیر بدیهی که از فیلوژنتیک محاسباتی استفاده میکند در نظر گرفته میشود. روشهای ماتریس فاصله از قبیل اتصال-همسایگی یا روش جفت گروه بدون وزن با میانگین حسابی که از روی دنباله تراز شده، فاصله ژنتیکی را محاسبه میکند، سادهترین روش برای پیادهسازی هستند، اما یک مدل تکاملی را احضار نمیکنند. هر روش تراز کردن دنباله از قبیل کلاستال از الگوریتم سادهتری (یعنی آنهایی که بر پایهٔ فاصله هستند) برای ساخت درخت استفاده میکند.
ماکسیمم پارسیمونی روش دیگری برای تخمین و محاسبه درخت فیلوژنتیک است، اما یک مدل ضمنی از تکامل ارائه میدهد (یعنی پارسیمونی).[۴] روشهای پیشرفتهتر، از معیار بهینگی ماکسیمم likelihood استفاده میکند. اغلب برپایهٔ بیزین، و یک مدل روشن از تکامل در تخمین درخت فیلوژنتیک، بهکار میبرد. مشخص کردن درخت بهینه با بسیاری از این روشها یک مسئله NP-سخت است.[۴] بنابراین جستجوی ابتکاری و روشهای بهینهسازی با توابع ارزشدهی درخت، جهت مشخصسازی یک درخت مناسب با دادهها، بهصورت ترکیبی استفاده میشوند.
روشهای ساخت درخت، میتواند بر اساس چند موضوع مورد بررسی قرار گیرد:[۶]
- مؤثر بودن: چه مقدار زمان نیاز دارد تا جواب را محاسبه کند؟ چه مقدار هزینه برای محاسبه جواب لازم دارد؟
- قدرت: از دادهها به خوبی استفاده میکند یا آنها را هدر میدهد؟
- سازگاری: آیا در صورت استفاده از دادههای مختلف برای یک مسئله به جوابهای یکسان همگرا خواهد شد؟
- پایداری: آیا بهخوبی از عهدهٔ فرضیات اشتباه بر میآید؟
جستارهای وابسته[ویرایش]
- درخت زندگی - راهی برای نشان دادن روابط میان ارگانیسمهای زنده و منقرضشده.
منابع[ویرایش]
- ↑ Hodge T, Cope M (1 October 2000). "A myosin family tree". J Cell Sci. 113 Pt 19 (19): 3353–4. PMID 10984423.
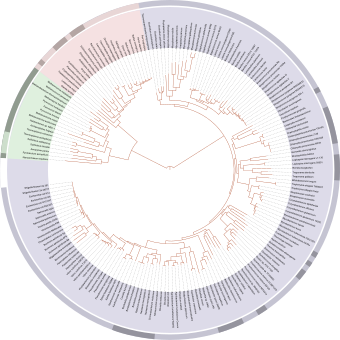
- ↑ Letunic, Ivica; Bork, Peer (2007-01-01). "Interactive Tree Of Life (iTOL): an online tool for phylogenetic tree display and annotation". Bioinformatics (Oxford, England). 23 (1): 127–128. doi:10.1093/bioinformatics/btl529. ISSN 1367-4811. PMID 17050570.
- ↑ Ciccarelli, Francesca D.; Doerks, Tobias; von Mering, Christian; Creevey, Christopher J.; Snel, Berend; Bork, Peer (2006-03-03). "Toward automatic reconstruction of a highly resolved tree of life". Science (New York, N.Y.). 311 (5765): 1283–1287. doi:10.1126/science.1123061. ISSN 1095-9203. PMID 16513982.
- ↑ ۴٫۰ ۴٫۱ ۴٫۲ Felsenstein J. (2004). Inferring Phylogenies Sinauer Associates: Sunderland, MA.
- ↑ Ali Asghar Geraili (۲۰۱۹-۰۸-۰۵). «ساختار درخت فیلوژنیک».[پیوند مرده]
- ↑ Penny, D. , Hendy, M. D. & M. A. Steel. 1992. Progress with methods for constructing evolutionary trees. Trends in Ecology and Evolution 7: 73-79.
برای مطالعهٔ بیشتر[ویرایش]
- Schuh, R. T. and A. V. Z. Brower. 2009. Biological Systematics: principles and applications (2nd edn.) ISBN 978-0-8014-4799-0
- MEGA, a free software to draw phylogenetic tress.
پیوند به بیرون[ویرایش]
| در ویکیانبار پروندههایی دربارهٔ Phylogenetic tree موجود است. |
تصاویر[ویرایش]
- درخت فیلوژنتیک برپایه 16 rDNA بایگانیشده در ۲۱ اوت ۲۰۰۸ توسط Wayback Machine
- تصویر با اندازه بزرگ از درخت حیات
- یک نمایش سه بعدی
- درخت کروموزومy در انسان -سال ۲۰۰۲
- درخت حیات
- درخت فیلوژنتیک جاندار مصنوعی شبیهسازی شده با کامپیوتر


 French
French Deutsch
Deutsch

