Exossoma – Wikipédia, a enciclopédia livre
O complexo do exossoma, complexo PM/Scl ou simplesmente exossoma é um complexo proteico multienzimático envolvido em diferentes passos do processamento e degradação de vários tipos de moléculas de ARN (ácido ribunocleico), através da sua atividade exonucleolítica. Os complexos do exossoma podem encontrar-se tanto em células eucariotas como nas arqueobactérias. Nas bactérias equivale a um complexo mais simples, o degradossoma, que desempenha funções similares.
O núcleo do complexo possui estrutura anular formada por seis membros, ao qual se acoplam outras proteínas. Nas células eucariotas encontra-se presente tanto no citoplasma como no núcleo celular, especialmente no nucléolo, embora as proteínas que interrelacionam com o complexo em cada um destes compartimentos sejam diferentes, regulando a sua atividade de degradação do ARN segundo a diferente natureza dos substratos presentes em cada compartimento celular.
Estes substratos compreendem o ARN mensageiro, ARN ribossomal e muitas espécies de pequenos ARNs bacterianos. O exossoma é constituído por até 11 subunidades que possuem atividade exorribonucleolítica, o que significa que degradam o ARN a partir de uma extremidade (chamada extremidade 3', neste caso) em vez de fragmentar o ARN em pontos específicos da molécula (como uma endonuclease faria).
Embora não haja relação causal entre o complexo e nenhuma das doenças conhecidas, algumas das suas proteínas são o antígeno de autoanticorpos responsáveis por algumas doenças autoimunes (especialmente pela síndrome de sobreposição de PM/Scl) e por algumas quimioterapias antimetabólicas que bloqueiam a actividade do complexo.
Descoberta[editar | editar código-fonte]
O exossoma foi identificado inicialmente como ribonuclease em 1997 na levedura Saccharomyces cerevisiae a partir da co-purificação de proteínas como Rrp4p e separação por gradiente de glicerol.[1] Não muito tempo depois, em 1999, viria a comprovar-se que o exossoma era, na verdade, o equivalente do complexo já descrito em células humanas, conhecido como complexo PM/Scl, que havia sido identificado nos anos anteriores como um autoantígeno em pacientes com certas doenças autoimunes (ver secção abaixo).[2] A purificação deste complexo humano permitiu a identificação de mais proteínas do exossoma e finalmente a caracterização de todos os seus componentes.[3][4] Em 2001 a crescente quantidade de dados procedentes dos distintos projetos genoma que se tornaram disponíveis permitiu a identificação de proteínas do exossoma de arqueas, ainda que tivesse transcorrido outros dois anos até que se identificasse o primeiro exossoma de um destes organismos.[5][6]
Estrutura[editar | editar código-fonte]
Proteínas centrais[editar | editar código-fonte]

O núcleo do complexo possui uma estrutura anular que consta de seis proteínas, todas elas pertencentes ao mesmo tipo de ribonuclease, as proteínas semelhantes às RNases PH.[7] Nas arqueas existem duas proteínas distintas deste tipo (chamadas Rrp41 e Rrp42), cada uma delas presente três vezes em ordem alternada. Os complexos de exossoma eucariota têm seis proteínas diferentes que formam a estrutura anular.[8][9] Dessas seis proteínas eucariotas, três semelham-se à proteína arqueota Rrp41 e as outras três apresentam maior semelhança com Rrp42.[10]
Situadas na parte superior do anel estão dispostas três proteínas que têm um domínio de união a ARN do tipo S1 (RBD). Duas proteínas, aliás, apresentam um domínio de homologia K (domínio KH).[7] Nas eucariotas unem-se três proteínas S1 diferentes ao anel, ao passo que nas arqueas podem fazer parte do exossoma um ou dois tipos destas proteínas (contudo, há sempre três subunidades S1 ligadas ao complexo).[11]

A estrutura em anel é muito similar à das proteínas RNases PH e polinucleótido fosforilases (PNPase). Nas bactérias, a RNase PH, envolvida no processamento do tRNA, forma um anel hexamérico consistente em seis RNases PH idênticas.[12] No caso da PNPase, que é uma proteína fosforolítica de degradação do ARN e que se encontra tanto em bactérias como em cloroplastos e mitocondrias dalguns organismos eucariotas, formam parte da mesma proteína dois domínios RNase PH e também um domínio S1 e outro de tipo KH de ligação a RNA. Esta proteína forma um complexo trimérico que adopta uma estrutura case idêntica à do exossoma.[13] Devido à grande similaridade entre os dois domínios de proteína e à sua estrutura, acredita-se que estes complexos estão relacionados evolutivamente e que têm um ancestral comum.[14] Nas bactérias existe uma RNase PH diferente que se encontra implicada no processamento do ARN de transferência, do qual é sabido que adopta uma estrutura similar anular de seis unidades, mas neste caso consiste de 6 subunidades proteicas idênticas.[15] As proteínas do exossoma semelhantes à RNase PH, às RNases PH e às PNPases pertencem todas elas à família das RNases e às exorribonucleases fosforolíticas, o que significa que utilizam fosfato inorgânico para eliminar nucleótidos do extremidade 3' das moléculas de ARN.[7]
Proteínas associadas[editar | editar código-fonte]
Para além das nove proteínas nucleares, outras duas proteínas são frequentemente associadas ao complexo de exossoma nos organismos eucariotas. Uma delas é Rrp44, uma RNase hidrolítica, pertencente à família RNase R de exorribonucleases hidrolíticas (nucleases que utilizam a água para dividir nucleótidos). Em leveduras, a Rrp44 está associada a todos os complexos de exossoma e apresenta um papel fundamental na atividade destes complexos.[16] Cabe referir que, apesar de haver uma proteína homóloga humana, não foi encontrada qualquer evidência de que esta esteja associada ao complexo de exossoma humano.[7]

A segunda proteína comum associada denomina-se Rrp6 (em leveduras) ou PM/Scl-100 (em humanos). Tal como a Rrp44, esta proteína também é uma exorribonuclease hidrolítica, porém neste caso pertence à família de proteínas RNase D.[17] A proteína PM/Scl-100 compreende a subunidade mais comum dos complexos de exossoma no núcleo das células, mas também constitui parte do complexo de exossoma citoplasmático.[18]
Proteínas reguladoras[editar | editar código-fonte]
Para além destas duas subunidades estritamente unidas, muitas proteínas interagem com o complexo exossómico tanto no citoplasma como no núcleo. Estas proteínas que se unem levemente podem regular a atividade e a especificidade do complexo de exossoma. No citoplasma, o exossoma interage com proteínas que se unem a elementos ricos em AU (ARE, por exemplo KRSP e TTP), que podem promover ou impedir a degradação dos ARNm. O exossoma nuclear associa-se às proteínas que ligam ARN (por exemplo MPP6 em humanos e Rrp47/C1D em humanos e leveduras) que controlam o processamento do ARN ribossomal.[7]
Além das proteínas individuais, outros complexos proteicos interagem com o exossoma. Um deles é o complexo Ski, que inclui uma ARN helicase (Ski2) e está relacionado com a degradação do ARNm.[19] No núcleo, o processamento do ARNr e snoRNA pelo exossoma é mediado pelo complexo TRAMP, que possui tanto a helicase de ARN Mtr4 como atividade de poliadenilação (Trf4).[20]
Função[editar | editar código-fonte]
Função enzimática[editar | editar código-fonte]
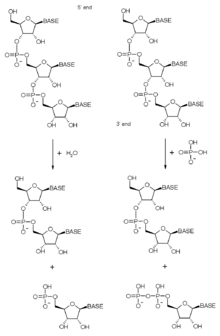
O complexo exossómico contém numerosas proteínas com domínios ribonuclease. Estes possuem atividade exorribonulcleasa 3'→ 5',[7] o que significa que as enzimas degradam as moléculas de ARN a partir da sua extremidade 3'. A natureza exata destes domínios de ribonuclease foi se alterando ao longo da evolução dos complexos bacterianos, arqueobacterianos e eucarioticos consoante o ganho e perda de diferentes actividades. As exorribonucleases contidas no exossoma podem ser fosforolíticas (proteínas de tipo RNAse PH) ou, em eucariotas, hidrolíticas (proteínas com domínios RNase D e R). As enzimas fosforolíticas utilizam fosfato inorgânico para romper as ligações fosfodiéster, libertando nucleosídeos difosfato,[21] e os hidrolíticos utilizam água para libertar nucleótidos (monofosfato).[22]
Em arqueotas, a subunidade Rrp41 do complexo é a exorribonuclease fosforolítica. O anel desta enzima apresenta três cópias desta proteína, que são as responsáveis pela atividade do complexo.[9] Em eucariotas, nenhuma das subunidades RNase PH terá mantido esta atividade catalítica, o que sugere que a estrutura anular principal do exossoma humano não tem nenhuma proteína enzimaticamente ativa.[23] Em leveduras isto é compensado por uma das enzimas hidrolíticas associadas, a Rrp44, que é a responsável pela maior parte da actividade ribonuclease do exossoma,[24] mas esta subunidade hidrolítica em particular pode estar restringida às leveduras, uma vez que não foi encontrada nenhuma proteína homóloga a Rrp44 no exossoma humano (e tão-pouco em arqueas).[25][26]
Podemos associar ainda outra enzima hidrolítica em humanos e leveduras com o complexo (Rrp6), que contribui para a atividade do exossoma de leveduras e é a única responsável pela atividade do complexo humano. Ainda que originalmente se pensasse que as proteínas com domínio S1 tinham também atividade hidrolítica 3'→5', pesquisas recentes têm posto em causa a existência desta atividade, ao afirmarem que estas proteínas poderiam desempenhar apenas um papel na ligação de substratos antes da sua degradação pelo complexo.[24]

Substratos[editar | editar código-fonte]
O exossoma está envolvido na degradação e processamento duma grande variedade de espécies de ARN. No citoplasma celular está relacionado com a renovação (turn-over) das moléculas de ARN mensageiro. O complexo pode degradar moléculas de ARNm que foram indicadas para a sua degradação por conter erros, a partir de interações com proteínas da via mediada por mutação sem sentido (nonsense) ou pela via mediada de mutação "non-stop". Por outro lado, os ARNm degradam-se como parte do seu ciclo normal. Existem várias proteínas que estabilizam ou desestabilizam moléculas de ARNm ligando-se aos elementos ricos em AU das extremidades 3' não traduzidos (3'UTR) e que interagem com o complexo do exossoma.[27][28][29]
No núcleo celular, o exossoma é necessário para o correcto processamento de vários pequenos RNAs nucleares (small nucleolar RNAs).[30] Por fim, o nucléolo é o compartimento onde se encontra a maior parte dos complexos do exossoma, onde exercem uma função no processamento do RNA ribossomal 5,8 S (a primeira função do exossoma identificada) e de vários RNA nucleolares pequenos.[1][30][31]
Ainda que a maior parte das células tenha outras enzimas com capacidade de degradar moléculas de ARN quer a partir da sua extremidade 3' quer a partir da sua extremidade 5', o exossoma é essencial para a sobrevivência celular. Quando a expressão génica das proteínas do exossoma é artificialmente diminuída ou bloqueada, por exemplo, através do ARN de interferência, o crescimento para e as células finalmente morrem. Tanto as proteínas centrais do complexo como as duas proteínas principais associadas são essenciais.[32]
As bactérias não possuem um complexo de exossoma; no entanto, as suas funções assumem um complexo similar, chamado degradossoma, através de um complexo simples que inclui a proteína polinucleótido fosforilase.[33]

Patologia[editar | editar código-fonte]
Autoimunidade[editar | editar código-fonte]
O complexo exossómico é o antígeno dos autoanticorpos responsáveis por diversas doenças autoimunes. Estes autoanticorpos encontram-se principalmente em pessoas que sofrem de síndrome de sobreposição de PM-Scl, uma doença autoimune na qual os pacientes sofrem uma esclerodermia de base acompanhada quer por polimiosite quer por dermatomiosite.[34] Os autoanticorpos podem ser detetados no soro sanguíneo dos pacientes a partir de vários ensaios. No passado, o método mais comum era o teste de imunodifusão dupla de Ouchterlony utilizando extratos de timo de vitelo, imunofluorescência em células HEp-2 ou imunoprecipitação a partir de extratos celulares humanos. Nos ensaios de imunoprecipitação com soros antiexossoma é precipitado um conjunto distinto de proteínas. Nos anos anteriores à identificação do exossoma, este padrão fora denominado de complexo PM-Scl.[35] Os soros destes pacientes normalmente apresentam uma coloração característica do nucléolo celular, alimentando a hipótese de que o antígeno reconhecido pelos autoanticorpos era importante para a síntese ribossomal.[36] Mais recentemente, tornou-se possível obter recombinantes de proteínas exossómicas, as quais têm sido utilizadas para desenvolver imunoanálises em linha (LIA) e ensaios de ELISA para detetar estes anticorpos.[7]
Nestas doenças, os anticorpos dirigem-se principalmente para duas das proteínas do complexo, denominadas PM/Scl-100 (a proteína similar a uma RNase D) e PM/Scl-75 (uma das proteínas similares às RNases PH do anel), e que são encontrados anticorpos que reconhecem estas proteínas em aproximadamente 30% dos pacientes que sofrem da síndrome.[37] Apesar de estas duas proteínas serem o ligando principal dos autoanticorpos, outras subunidades ou proteínas associadas podem também ser orientadas nestes pacientes, como é exemplo o C1D.[38][39] Atualmente, o método mais sensível para detetar estes autoanticorpos consiste na utilização dum péptido derivado da proteína PM/Scl-100 como antígeno num teste ELISA, ao invés de utilizar proteínas completas. Através deste método, os autoanticorpos encontram-se em mais de 55% dos pacientes com a síndrome de sobreposição, embora possam também ser detectados em pacientes que sofrem tanto de esclerodermia, como de polimiosite ou de dermatomiosite, à parte.[40]
Dado que os autoanticorpos se encontram principalmente em pacientes que possuem características de muitas doenças autoimunes distintas, os sintomas clínicos que apresentam podem variar significativamente. Os sintomas que se vêem com mais frequência são os sintomas típicos de doenças autoimunes individuais, entre elas o signo de Raynaud, artrite, miosite e esclerodermia.[41] O tratamento destes pacientes é sintomático e assemelha-se ao tratamento de cada uma das doença autoimunes particular, o que envolve a administração de fármacos imunosupressores ou imunomoduladores.[42]
Cancro[editar | editar código-fonte]
Foi demonstrado que o exossoma é inibido pelo antimetabolito 5-fluorouracilo, utilizado também no tratamento quimioterapéutico do cancro. Este é um dos tratamentos mais eficazes contra os tumores sólidos. Quando este fármaco é administrado a leveduras, podem-se encontrar defeitos no processamento do ARN ribossomal idênticos aos observados quando se bloqueia a atividade do exossoma através de diferentes métodos utilizados em biologia molecular. A ausência de um correto processamento dos ribossomas é letal para estas células, o que explica o seu efeito antimetabólico.[43]
Lista de subunidades[editar | editar código-fonte]
| Legenda | Nome geral | Domínios | Homo sapiens | S. cerevisiae | Archaea | Massa molecular (kDa) | Gene humano | Gene de levedura |
|---|---|---|---|---|---|---|---|---|
| 1 | Csl4 | S1 RBD | hCsl4 | Csl4p/Ski4p | Csl4 | 21-32 | EXOSC1 | YNL232W |
| 2 | Rrp4 | S1/KH RBD | hRrp4 | Rrp4p | Rrp4 | 28-39 | EXOSC2 | YHR069C |
| 3 | Rrp40 | S1/KH RBD | hRrp40 | Rrp40p | (Rrp4)A | 27-32 | EXOSC3 | YOL142W |
| 4 | Rrp41 | RNase PH | hRrp41 | Rrp41p/Ski6p | Rrp41 | 26-28 | EXOSC4 | YGR195W |
| 5 | Rrp46 | RNase PH | hRrp46 | Rrp46p | (Rrp41)A | 25-28 | EXOSC5 | YGR095C |
| 6 | Mtr3 | RNase PH PH | hMtr3 | Mtr3p | (Rrp41)A | 24-37 | EXOSC6 | YGR158C |
| 7 | Rrp42 | RNase PH | hRrp42 | Rrp42p | Rrp42 | 29-32 | EXOSC7 | YDL111C |
| 8 | Rrp43 | RNase PH | OIP2 | Rrp43p | (Rrp42)A | 30-44 | EXOSC8 | YCR035C |
| 9 | Rrp45 | RNase PH | PM/Scl-75 | Rrp45p | (Rrp42)A | 34-49 | EXOSC9 | YDR280W |
| 10 | Rrp6 | RNase D | PM/Scl-100 | Rrp6p | n/a | 84-100 | EXOSC10 | YOR001W |
| 11 | Rrp44 | RNase R | (hDis3)B | Rrp44p/Dis3p | n/a | 105-113 | KIAA1008 | YOL021C |
- A : Nas arqueobactérias várias proteínas do exossoma encontram-se presentes em múltiplas cópias para formar o núcleo completo do complexo exossómico.
- B : Apesar de a proteína Rrp44 ser parte do complexo na levedura, o seu homólogo humano, hDis3, não nunca foi encontrado associado ao complexo humano.
- C : A proteína humana PM/Scl-100 contribui para a atividade ribonucleolítica do complexo.
Ver também[editar | editar código-fonte]
- Proteassoma, o principal complexo celular de degradação de proteínas.
- Spliceossoma, um complexo relacionado com o processo denominado splicing de ARNm, que também possui uma estrutura anular de ligação ao RNA.
- Vault (organela), uma organela ribonucleoproteica.
Referências
- ↑ a b Mitchell; et al. (1997). «The Exosome: A Conserved Eukaryotic ARN Processing Complex Containing Multiple 3′→5′ Exoribonucleases». Cell. 91 (4): 457–466. PMID 9390555. doi:10.1016/S0092-8674(00)80432-8
- ↑ Allmang; et al. (1999). «The yeast exosome and human PM-Scl are related complexes of 3'→ 5' exonucleases». Genes and Development. 13 (16): 2148–58. PMID 10465791. doi:10.1101/gad.13.16.2148
- ↑ Brouwer; et al. (2001). «Three novel components of the human exosome». Journal of Biological Chemistry. 276: 6177–84. PMID 11110791. doi:10.1074/jbc.M007603200
- ↑ Chen; et al. (2001). «AU binding proteins recruit the exosome to degrade ARE-containing mARNs». Cell. 107: 451–64. PMID 11719186. doi:10.1016/S0092-8674(01)00578-5
- ↑ Koonin; et al. (2001). «Prediction of the archaeal exosome and its connections with the proteasome and the translation and transcription machineries by a comparative-genomic approach». Genome Research. 11 (2): 240–52. PMID 11157787. doi:10.1101/gr.162001
- ↑ Evguenieva-Hackenberg; et al. (2003). «An exosome-like complex in Sulfolobus solfataricus». EMBO Reports. 4 (9): 889–93. PMID 12947419. doi:10.1038/sj.embor.embor929
- ↑ a b c d e f g Schilders; et al. (2006). «Cell and molecular biology of the exosome: how to make or break an RNA». International review of cytology. 251: 159–208. PMID 16939780. doi:10.1016/S0074-7696(06)51005-8
- ↑ Lorentzen; et al. (2005). «The archaeal exosome core is a hexameric ring structure with three catalytic subunits». Nature Structural & Molecular Biology. 12: 575–81. PMID 15951817. doi:10.1038/nsmb952
- ↑ a b Shen; et al. (2006). «A view to a kill: structure of the RNA exosome». Cell. 127: 1093–5. PMID 17174886. doi:10.1016/j.cell.2006.11.035
- ↑ Raijmakers; et al. (2002). «Protein-protein interactions between human exosome components support the assembly of RNase PH-type subunits into a six-membered PNPase-like ring». Journal of Molecular Biology. 323: 653–63. PMID 12419256. doi:10.1016/S0022-2836(02)00947-6
- ↑ Walter; et al. (2006). «Characterization of native and reconstituted exosome complexes from the hyperthermophilic archaeon Sulfolobus solfataricus». Molecular Microbiology. 62: 1076–89. PMID 17078816. doi:10.1111/j.1365-2958.2006.05393.x
- ↑ Ishii; et al. (2003). «Crystal structure of the ARNt processing enzyme RNase PH from Aquifex aeolicus». Journal of Biological Chemistry. 278: 32397–404. PMID 12746447. doi:10.1074/jbc.M300639200
- ↑ Symmons; et al. (2000). «A duplicated fold is the structural basis for polynucleotide phosphorylase catalytic activity, processivity, and regulation». Structure. 8: 1215–26. PMID 11080643. doi:10.1016/S0969-2126(00)00521-9
- ↑ Lin-Chao; et al. (2007). «The PNPase, exosome and RNA helicases as the building components of evolutionarily-conserved RNA degradation machines». Journal of Biomedical Science. 14: 523–32. doi:10.1007/s11373-007-9178-y
- ↑ Harlow; et al. (2004). «Crystal structure of the phosphorolytic exoribonuclease RNase PH from Bacillus subtilis and implications for its quaternary structure and tRNA binding». Protein Science. 13: 668–77. PMID 14767080. doi:10.1110/ps.03477004
- ↑ Schneider; et al. (2007). «The exosome subunit Rrp44 plays a direct role in ARN substrate recognition». Molecular Cell. 27: 324–31. PMID 17643380. doi:10.1016/j.molcel.2007.06.006
- ↑ Mian; et al. (1997). «Comparative sequence analysis of ribonucleases HII, III, II PH and D». Nucleic Acids Research. 25: 3187–3195. PMID 9241229. doi:10.1093/nar/25.16.3187
- ↑ Raijmakers; et al. (2004). «The exosome, a molecular machine for controlled RNA degradation in both nucleus and cytoplasm». European Journal of Cell Biology. 83: 175–83. PMID 15346807. doi:10.1078/0171-9335-00385
- ↑ Wang; et al. (2005). «Domain interactions within the Ski2/3/8 complex and between the Ski complex and Ski7p». RNA. 11: 1291–302. PMID 16043509. doi:10.1261/rna.2060405
- ↑ LaCava; et al. (2005). «RNA degradation by the exosome is promoted by a nuclear polyadenylation complex». Cell. 121: 713–24. PMID 15935758. doi:10.1016/j.cell.2005.04.029
- ↑ «Exoribonuclease, phosphorolytic domain 2». InterPro Protein Domain record. Riken Meta Database. Consultado em 8 de dezembro de 2015
- ↑ Torben Heick Jensen (2011). RNA Exosome. [S.l.: s.n.] p. 19. ISBN 1441978410
- ↑ Liu; et al. (2007). «Erratum: Reconstitution, activities, and structure of the eukaryotic RNA exosome». Cell. 131: 188–189. PMID 17174896. doi:10.1016/j.cell.2007.09.019
- ↑ a b Dziembowski; et al. (2007). «A single subunit, Dis3, is essentially responsible for yeast exosome core activity». Nature Structural & Molecular Biology. 14: 15–22. PMID 17173052. doi:10.1038/nsmb1184
- ↑ Liu; et al. (2006). «Reconstitution, activities, and structure of the eukaryotic RNA exosome». Cell. 127: 1223–37. PMID 1717489. doi:10.1016/j.cell.2006.10.037
- ↑ Lorentzen; et al. (2005). «Structural basis of 3' end RNA recognition and exoribonucleolytic cleavage by an exosome RNase PH core». Molecular Cell. 20: 473–81. PMID 16285928. doi:10.1016/j.molcel.2005.10.020
- ↑ LeJeune; et al. (2003). «Nonsense-mediated mRNA decay in mammalian cells involves decapping, deadenylating, and exonucleolytic activities». Molecular Cell. 12: 675–87. PMID 14527413. doi:10.1016/S1097-2765(03)00349-6
- ↑ Wilson; et al. (2007). «A genomic screen in yeast reveals novel aspects of nonstop mRNA metabolism». Genetics. 177. 773 páginas. PMID 17660569. doi:10.1534/genetics.107.073205
- ↑ Lin; et al. (2007). «Localization of AU-rich element-containing mRNA in cytoplasmic granules containing exosome subunits». Journal of Biological Chemistry. 282: 19958–68. PMID 17470429. doi:10.1074/jbc.M702281200
- ↑ a b Allmang; et al. (1999). «Functions of the exosome in rRNA, snoRNA and snRNA synthesis». EMBO Journal. 18: 5399–410. PMID 10508172. doi:10.1093/emboj/18.19.5399
- ↑ Schilders; et al. (2005). «MPP6 is an exosome-associated RNA-binding protein involved in 5.8S rRNA maturation». Nucleic Acids Research. 33: 6795–804. PMID 16396833. doi:10.1093/nar/gki982
- ↑ van Dijk; et al. (2007). «Human cell growth requires a functional cytoplasmic exosome, which is involved in various mRNA decay pathways». RNA. 13: 1027–35. PMID 17545563. doi:10.1261/rna.575107
- ↑ Carpousis AJ (2002). «The Escherichia coli RNA degradosome: structure, function and relationship in other ribonucleolytic multienzyme complexes». Biochem. Soc. Trans. 30 (2): 150–5. PMID 12035760. doi:10.1042/BST0300150
- ↑ J.E. Pope (2002). «Scleroderma overlap syndromes». Current Opinion in Rheumatology. 14: 704–10. PMID 12410095. doi:10.1097/00002281-200211000-00013
- ↑ Gelpi; et al. (1991). «Identification of protein components reactive with anti-PM/Scl autoantibodies». Clinical and Experimental Immunology. 81: 59–64. PMID 2199097
- ↑ Targoff; et al. (1985). «Nucleolar localization of the PM-Scl antigen». Arthritis & Rheumatism. 28: 226–30. PMID 3918546. doi:10.1002/art.1780280221
- ↑ Raijmakers; et al. (2004). «PM-Scl-75 is the main autoantigen in patients with the polymyositis/scleroderma overlap syndrome». Arthritis & Rheumatism. 50: 565–9. PMID 14872500. doi:10.1002/art.20056
- ↑ Brouwer; et al. (2002). «Autoantibodies directed to novel components of the PM/Scl complex, the human exosome.». Arthritis Research. 4: 134–8. PMID 11879549. doi:10.1186/ar389
- ↑ Schilders; et al. (2007). «C1D is a major autoantibody target in patients with the polymyositis-scleroderma overlap syndrome». Arthritis & Rheumatism. 56: 2449–54. PMID 17599775. doi:10.1002/art.22710
- ↑ Mahler; et al. (2005). «Clinical evaluation of autoantibodies to a novel PM/Scl peptide antigen». Arthritis Research & Therapy. 7: R704–13. PMID 15899056. doi:10.1186/ar1729
- ↑ Mahler; et al. (2007). «Novel aspects of autoantibodies to the PM/Scl complex: Clinical, genetic and diagnostic insights». Autoimmunity Reviews. 6: 432–7. PMID 17643929. doi:10.1016/j.autrev.2007.01.013
- ↑ Jablonska; et al. (1998). «Scleromyositis: a scleroderma/polymyositis overlap syndrome». Clinical Rheumatology. 17: 465–7. PMID 9890673. doi:10.1007/BF01451281
- ↑ Lum; et al. (2004). «Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes». Cell. 116: 121–37. PMID 14718172. doi:10.1016/S0092-8674(03)01035-3
Bibliografia[editar | editar código-fonte]
- Schilders, G; Pruijn, GJ (2008). «Biochemical studies of the mammalian exosome with intact cells». Methods Enzymol. Methods in Enzymology. 448: 211–226. ISBN 9780123743787. PMID 19111178. doi:10.1016/S0076-6879(08)02611-6
- Houseley, J; Tollervey, D (2008). «The nuclear RNA surveillance machinery: the link between ncRNAs and genome structure in budding yeast?». Biochim Biophys Acta. 1779 (4): 239–246. PMID 18211833. doi:10.1016/j.bbagrm.2007.12.008
- Vanacova, S; Stefl, R (2007). «The exosome and RNA quality control in the nucleus». EMBO Reports. 8 (7): 651–657. PMC 1905902
 . PMID 17603538. doi:10.1038/sj.embor.7401005
. PMID 17603538. doi:10.1038/sj.embor.7401005 - Houseley, J; Lacava, J; Tollervey, D (2006). «RNA-quality control by the exosome». Nature Reviews Molecular Cell Biology. 7 (7): 529–539. PMID 16829983. doi:10.1038/nrm1964 –-- subscription required
- Büttner, K; Wenig, K; Hopfner, KP (2006). «The exosome: a macromolecular cage for controlled RNA degradation». Molecular Microbiology. 61 (6): 1372–1379. PMID 16968219. doi:10.1111/j.1365-2958.2006.05331.x
- Lorentzen, E; Conti, E (2006). «The Exosome and the Proteasome: Nano-Compartments for Degradation». Cell. 125 (4): 651–654. PMID 16713559. doi:10.1016/j.cell.2006.05.002
- Pruijn, GJ (2005). «Doughnuts dealing with RNA». Nature Structural & Molecular Biology. 12 (7): 562–564. PMID 15999107. doi:10.1038/nsmb0705-562
Ligações externas[editar | editar código-fonte]
- Estrutura do exossoma humano em RCSB Protein Data Bank
- Estrutura de um exossoma de arquea em RCSB Protein Data Bank
- Estrutura da proteína do exosoma de levedura Rrp6 en RCSB Protein Data Bank


 French
French Deutsch
Deutsch