Identidade por descendência – Wikipédia, a enciclopédia livre
Um segmento de DNA é idêntico por estado (IPE) em dois ou mais indivíduos se eles tiverem sequências de nucleotídeos idênticas neste segmento. Um segmento IPE é idêntico por descendência (IPD) em dois ou mais indivíduos se o tiverem herdado de um ancestral comum sem recombinação, ou seja, o segmento tem a mesma origem ancestral nestes indivíduos. Os segmentos de DNA que são IPD são IPE por definição, mas os segmentos que não são IPD ainda podem ser IPE devido às mesmas mutações em diferentes indivíduos ou recombinações que não alteram o segmento.

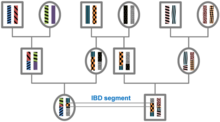
Teoria[editar | editar código-fonte]
Todos os indivíduos numa população finita estão relacionados se forem rastreados há tempo suficiente e irão, portanto, partilhar segmentos dos seus genomas com IPD. Durante a meiose, segmentos da IPD são quebrados por recombinação. Portanto, o comprimento esperado de um segmento IPD depende do número de gerações desde o ancestral comum mais recente no locus do segmento. O comprimento dos segmentos de IPD que resultam de um ancestral comum n gerações no passado (envolvendo, portanto, 2n meiose) é distribuído exponencialmente com média 1/(2n) Morgans (M). [1] O número esperado de segmentos IPD diminui com o número de gerações desde o ancestral comum neste locus. Para um segmento específico de DNA, a probabilidade de ser IPD diminui em 2−2n, pois em cada meiose a probabilidade de transmitir esse segmento é 1/2. [2]
Usos[editar | editar código-fonte]
Os segmentos IPD identificados podem ser usados para uma ampla gama de finalidades. Conforme observado acima, a quantidade (comprimento e número) de compartilhamento de IPD depende das relações familiares entre os indivíduos testados. Portanto, uma aplicação da detecção do segmento IPD é quantificar o parentesco. [3] [4] [5] [6] A medição do parentesco pode ser usada em genética forense, [7] mas também pode aumentar as informações no mapeamento de ligação genética [3] [8] e ajudar a diminuir o viés por relacionamentos não documentados em estudos de associação padrão. [6] [9] Outra aplicação do IPD é a imputação de genótipos e a inferência de fases de haplótipos. [10] [11] [12] Longos segmentos partilhados de IPD, que são divididos por regiões curtas, podem ser indicativos de erros de faseamento. [5] [13]
Veja também[editar | editar código-fonte]
- Ligação genética
- Estudo de associação em todo o genoma
- Desequilíbrio de ligação
- Genética populacional
Referências[editar | editar código-fonte]
- ↑ Browning, S. R. (2008). «Estimation of Pairwise Identity by Descent from Dense Genetic Marker Data in a Population Sample of Haplotypes». Genetics. 178 (4): 2123–2132. PMC 2323802
 . PMID 18430938. doi:10.1534/genetics.107.084624
. PMID 18430938. doi:10.1534/genetics.107.084624 - ↑ Thompson, E. A. (2008). «The IBD process along four chromosomes». Theoretical Population Biology. 73 (3): 369–373. PMC 2518088
 . PMID 18282591. doi:10.1016/j.tpb.2007.11.011
. PMID 18282591. doi:10.1016/j.tpb.2007.11.011 - ↑ a b Albrechtsen, A.; Sand Korneliussen, T.; Moltke, I.; Van Overseem Hansen, T.; Nielsen, F. C.; Nielsen, R. (2009). «Relatedness mapping and tracts of relatedness for genome-wide data in the presence of linkage disequilibrium». Genetic Epidemiology. 33 (3): 266–274. PMID 19025785. doi:10.1002/gepi.20378
- ↑ Browning, S. R.; Browning, B. L. (2010). «High-Resolution Detection of Identity by Descent in Unrelated Individuals». The American Journal of Human Genetics. 86 (4): 526–539. PMC 2850444
 . PMID 20303063. doi:10.1016/j.ajhg.2010.02.021
. PMID 20303063. doi:10.1016/j.ajhg.2010.02.021 - ↑ a b Gusev, A.; Lowe, J. K.; Stoffel, M.; Daly, M. J.; Altshuler, D.; Breslow, J. L.; Friedman, J. M.; Pe'Er, I. (2008). «Whole population, genome-wide mapping of hidden relatedness». Genome Research. 19 (2): 318–326. PMC 2652213
 . PMID 18971310. doi:10.1101/gr.081398.108
. PMID 18971310. doi:10.1101/gr.081398.108 - ↑ a b Purcell, S.; Neale, B.; Todd-Brown, K.; Thomas, L.; Ferreira, M. A. R.; Bender, D.; Maller, J.; Sklar, P.; De Bakker, P. I. W. (2007). «PLINK: A Tool Set for Whole-Genome Association and Population-Based Linkage Analyses». The American Journal of Human Genetics. 81 (3): 559–575. PMC 1950838
 . PMID 17701901. doi:10.1086/519795
. PMID 17701901. doi:10.1086/519795 - ↑ Ian W. Evett; Bruce S. Weir (janeiro de 1998). Interpreting DNA Evidence: Statistical Genetics for Forensic Scientists. [S.l.]: Sinauer Associates, Incorporated. ISBN 978-0-87893-155-2
- ↑ Leutenegger, A.; Prum, B.; Genin, E.; Verny, C.; Lemainque, A.; Clergetdarpoux, F.; Thompson, E. (2003). «Estimation of the Inbreeding Coefficient through Use of Genomic Data». The American Journal of Human Genetics. 73 (3): 516–523. PMC 1180677
 . PMID 12900793. doi:10.1086/378207
. PMID 12900793. doi:10.1086/378207 - ↑ Voight, B. F.; Pritchard, J. K. (2005). «Confounding from Cryptic Relatedness in Case-Control Association Studies». PLOS Genetics. 1 (3): e32. PMC 1200427
 . PMID 16151517. doi:10.1371/journal.pgen.0010032
. PMID 16151517. doi:10.1371/journal.pgen.0010032 - ↑ Kong, A.; Masson, G.; Frigge, M. L.; Gylfason, A.; Zusmanovich, P.; Thorleifsson, G.; Olason, P. I.; Ingason, A.; Steinberg, S. (2008). «Detection of sharing by descent, long-range phasing and haplotype imputation». Nature Genetics. 40 (9): 1068–1075. PMC 4540081
 . PMID 19165921. doi:10.1038/ng.216
. PMID 19165921. doi:10.1038/ng.216 - ↑ Gusev, A.; Shah, M. J.; Kenny, E. E.; Ramachandran, A.; Lowe, J. K.; Salit, J.; Lee, C. C.; Levandowsky, E. C.; Weaver, T. N. (2011). «Low-Pass Genome-Wide Sequencing and Variant Inference Using Identity-by-Descent in an Isolated Human Population». Genetics. 190 (2): 679–689. PMC 3276614
 . PMID 22135348. doi:10.1534/genetics.111.134874
. PMID 22135348. doi:10.1534/genetics.111.134874 - ↑ Browning, B. L.; Browning, S. R. (2009). «A Unified Approach to Genotype Imputation and Haplotype-Phase Inference for Large Data Sets of Trios and Unrelated Individuals». The American Journal of Human Genetics. 84 (2): 210–223. PMC 2668004
 . PMID 19200528. doi:10.1016/j.ajhg.2009.01.005
. PMID 19200528. doi:10.1016/j.ajhg.2009.01.005 - ↑ Hochreiter, S. (2013). «HapFABIA: Identification of very short segments of identity by descent characterized by rare variants in large sequencing data». Nucleic Acids Research. 41 (22): e202. PMC 3905877
 . PMID 24174545. doi:10.1093/nar/gkt1013
. PMID 24174545. doi:10.1093/nar/gkt1013


 French
French Deutsch
Deutsch